为了抵抗病原菌的侵染, 植物在长久的进化过程中形成了一系列的防御机制, 其中系统获得抗性(systemic acquired resistance, SAR)是一种可诱导的植物抗性反应, 并能够提供持久的广谱性抗病作 用[1]。SAR反应的诱发需要水杨酸(salicylic acid, SA)的积累, 并且伴随着病程相关蛋白(pathogenesis- related protein)的协同表达[2]。 NPR1( nonexpressor of pathogenesis related genes 1)基因是SA介导的SAR反应中的核心调控元件[3,4], 该基因编码的蛋白包含一个位于N端BTB/POZ (broad-complex, tramtrack, and bric-a-brac/pox virus and zinc finger)结构域和一个位于中间区域锚蛋白重复结构域ANK (ankyin repeat domain), 这2个结构域在蛋白质间的相互作用中起重要作用[5,6]。在未受到病原菌侵袭时, NPR1蛋白通过二硫键相互结合, 以多聚体的形式存在于细胞质中。当感染病原菌后, SA的积累改变了细胞中的氧化还原势, 导致NPR1单体的释放。NPR1单体进入细胞核后与TGA-bZIP转录因子结合, 进而激活防御反应相关基因的表达, 最终引起SAR反 应[7,8]。研究表明, 依赖于茉莉酸(jasmonic acid, JA)和乙烯(ethylene, ET)介导的诱导系统抗性(inducted systemic resistance, ISR)反应同样也需要 NPR1的参与[9]。因此, 作为必需的调控基因, NPR1参与调控SAR与ISR反应并在整个抗病网络中发挥重要作用。目前, 已从拟南芥[5]、水稻[10]、心叶烟[11]、中间偃麦草[12]、棉花[13]、苹果[14]等多种植物中分离到 NPR1基因及同源基因, 并对部分基因的功能进行了不同程度的研究。在转基因水稻中过表达 AtNPR1和水稻 NPR1同源基因均明显提高对白叶枯病的抗性[15]。苹果植株转 MpNPR1基因后提高了对苹果黑星病菌和桧胶锈菌的抗性[14]。在拟南芥中过表达 AtNPR1基因, 增强了对丁香假单胞菌和寄生霜霉菌的抗性[16], 而将 NPR1基因突变后, 突变植株更容易受到这些病原菌的侵染[5]。
赤霉病是由禾谷镰刀菌( Fusarium graminearum)引起的一种严重的小麦穗部病害, 除造成产量的降低及经济损失外, 在侵染过程中还产生多种真菌毒素, 严重危害人类和动物的健康[17]。尽管对赤霉病的研究已经做了大量工作, 但其抗病机制尚不明确。Makandar等[18]把拟南芥 AtNPR1基因转入小麦品种Bobwhite中, 提高了转基因植株对赤霉菌的抗性。目前, 在小麦上仅检索到一次 NPR1同源基因 NPH2( NPR1homolog 2)的专利注册(专利号: WO0070069), 而未见对小麦 NPR1的研究报道。在
拟南芥、水稻、玉米、短柄草等植物中均发现多个 NPR1同源序列, 而且研究表明在拟南芥中, NPR1的同源物NPR3和NPR4对NPR1介导的抗病反应起到重要的调控作用[19]。小麦是异源六倍体, 基因组庞大, 可能存在更多的 NPR1同源基因。因此, 克隆并研究小麦 NPR1及其同源基因, 对于揭示小麦与赤霉菌的相互作用并探讨小麦赤霉病抗病机制具有十分重要的意义。本研究从小麦中克隆了3个 NPR1-like基因 TaNPR1、 TaNPR2和 TaNPR3的全长cDNA序列, 并进行序列分析和功能预测, 同时还对SA、茉莉酸甲酯(MeJA)及赤霉菌诱导下这3个基因的表达特性进行了研究。
小麦抗、感赤霉病近等基因系Apogee73S2 (抗)和Apogee (感)由美国明尼苏达大学D. Garvin博士提供。其创制过程是, 以抗赤霉病小麦品种苏麦3号为供体亲本, 感病材料Apogee为轮回亲本, 回交4代后自交得到基因型近于纯合的抗赤霉病近等基因系Apogee73S2。在回交和自交过程中, 利用与 Fhb1紧密连锁的分子标记 Xgwm493和 Xgwm533对目标基因逐代进行分子辅助筛选, 最后选育出抗、感赤霉病近等基因系。
将2个近等基因系小麦幼苗在4℃培养箱中春化3周, 然后转移到温室中, 每天光照16 h, 平均温度28℃(昼)/16℃(夜)。以绿豆汤做培养基, 25℃振荡培养禾谷镰刀菌, 使其产生孢子。用无菌水悬浮孢子, 使菌液浓度为1×106mL-1。在开花期以单花接种法接种禾谷镰刀菌( F. graminearum), 每小花接种10 μL菌液, 以无菌水处理作对照。接种后用透明塑料袋套袋保湿, 在接种后12、24、48和72 h采集颖壳, 每个时间点分别取10穗。禾谷镰刀菌( F. graminearum)由南京农业大学王秀娥教授提供。
以抗病系Apogee73S2为实验材料, 分别用SA和MeJA (均为2 mmol L-1)浸泡萌发后1周的小麦幼苗根部, 于诱导处理0 (对照)、1、2、4、8、12和24 h后取样(根上部)。
所有样品采集后立即用液氮处理并置-80℃保存。采用TIANGEN RNA试剂盒提取上述实验材 料的总RNA, 再利用TransGen试剂盒合成cDNA第一链。
采集赤霉菌处理72 h及对照处理的颖壳, 提取其总RNA构建差异表达谱, 从中获得3条小麦 NPR1及其同源基因的EST片段, 然后查询Triticeae Full-Length CDS Database (http://trifldb.psc.riken.jp/ index.pl)得到其cDNA序列。根据cDNA序列设计引物(表1), 以接菌后72 h的cDNA为模板进行扩增。PCR扩增体系为20 μL, 含10× Taq buffer (Mg2+) 2 μL、模板2 μL、10 μmol L-1上下游引物各0.5 μL、2.5 mmol L-1 dNTPs 1.5 μL、5 U L-1 Taq DNA聚合酶0.3 μL、H2O 13.2 μL。扩增程序为94℃ 5 min; 94℃ 30 s, 60℃ 30 s, 72℃ 2 min, 36个循环; 72℃延伸10 min。反应产物经1%的琼脂糖凝胶电泳分离, 然后进行目的带回收、克隆和测序。
利用NCBI的在线分析工具ORF Finder预测基因的开放阅读框; 用ProtParam (http://expasy.org/ tools/protparam.html)在线分析氨基酸序列的理化性质; 利用SOPMA对编码蛋白的二级结构进行预测; 利用在线工具Pfam (http://pfam.sanger.ac.uk/)预测基因编码蛋白的结构域; 用DNAMAN和MEGA 4.0软件进行相关蛋白序列的同源性比对, 构建进 化树。
根据 TaNPR1、 TaNPR2和 TaNPR3基因的序列, 设计特异的荧光定量PCR引物(表1), 以SA、MeJA处理的幼苗cDNA及赤霉菌和无菌水处理的颖壳cDNA为模板, 利用Bio-Rad CFX96TM实时荧光定量PCR仪检测目的基因表达模式。内参基因为 β-Actin[20], 其引物序列见表1。按照SYBR Primix Ex Taq试剂盒(TaKaRa)说明书配制PCR反应体系。反
应条件为95℃ 30 s; 95℃ 10 s, 57℃ 15 s, 72℃ 20 s, 40个循环。采用2-ΔΔCT法对目的基因进行相对定量表达分析。每个样品设3次重复, 每次处理有3次生物学重复。用DPS软件作差异显著性分析(LSD法)。
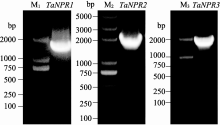
通过RT-PCR对 TaNPR1、 TaNPR2和 TaNPR3的全长cDNA进行扩增, PCR产物经1%琼脂糖凝胶电泳后分别得到1775、1848和1827 bp的目的带(图1)。测序结果显示, TaNPR1开放阅读框长度为1743 bp, 预测编码580个氨基酸, 蛋白分子量为63.6 kD, 理论等电点p I为5.22, 而在小麦数据库Triticeae Full-Length CDS Database中比对到的cDNA全长仅能编码473个氨基酸, 因此克隆得到的 TaNPR1为一个新基因。 TaNPR2与 TaNPR3的开放阅读框长度分别为1824 bp和1806 bp, 编码607个和601个氨基酸, 两者的蛋白分子量较为接近, 但等电点相差较大, 分别为5.27和5.70 (表2)。TaNPR1与TaNPR2和TaNPR3的氨基酸序列相似度分别为41%和40%, 而将小麦NPR1同源物NPH2与TaNPR1、TaNPR2、TaNPR3比较, 发现其与TaNPR2序列十分相似, 相似度为97%, 但与TaNPR1、TaNPR3相似度仅为41%和52%。对氨基酸的理化性质及蛋白二级结 构预测分析后发现(表2), 3个小麦NPR1-like蛋白 均为脂溶性的疏水蛋白, 其二级结构中都包含α 螺旋、延伸链、β折叠和无规则卷曲。TaNPR1和TaNPR2均含有69个碱性氨基酸, 而TaNPR3在酸性和碱性氨基酸数目上均高于TaNPR1与TaNPR2 (表2)。
| 表1 TaNPR1、 TaNPR2和 TaNPR3克隆及表达分析所用引物 Table 1 Primers for TaNPR1, TaNPR2, and TaNPR3isolation and qRT-PCR analysis |
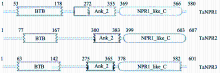
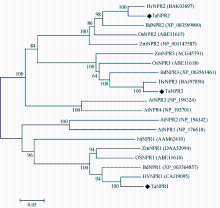
通过Pfam (http://pfam.sanger.ac.uk/)以及NCBI的BLASTP对TaNPR1、TaNPR2和TaNPR3的蛋白结构域及序列相似性分析表明TaNPR1、TaNPR2和TaNPR3均含有与AtNPR1类似的BTB/POZ、ANK和NPR1_like_C结构域(图2)。TaNPR1与拟南芥AtNPR1蛋白的同源性最高, 为46%, 而TaNPR2和TaNPR3与AtNPR1的蛋白同源性则分别为38%和40%。对小麦、拟南芥、烟草、水稻的NPR1及NPR1同源蛋白的氨基酸序列的多重比对分析发现, TaNPR1、TaNPR2和TaNPR3均含有保守的npr1-1、npr1-2、nim1-4功能位点(图3), 这些氨基酸残基对于NPR1功能的行使起重要作用。此外, TaNPR1还具有对NPR1寡聚体形成所必需的2个保守半胱氨酸残基, 分别为Cys71和Cys211。不同物种NPR1及NPR1同源蛋白的进化树表明, NPR1蛋白与NPR1同源蛋白在进化上形成不同分支, TaNPR1和TaNPR2、TaNPR3同源性较低。TaNPR1、TaNPR2、TaNPR3分别与大麦、短柄草、水稻、玉米的NPR1、NPR2及NPR3蛋白聚为一类, 并与大麦的NPR蛋白同源关系最近(图4)。
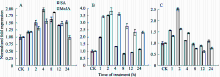
为了检测 TaNPR1、 TaNPR2和 TaNPR3对抗病信号分子的响应情况, 分别用SA和MeJA对抗赤霉病近等基因系Apogee73S2进行处理。SA和MeJA处理后, TaNPR1表达量升高, 分别在处理4 h和8 h后出现表达高峰, 与各自对照相比均差异显著( P<0.05)。与 TaNPR1相比, TaNPR2和 TaNPR3对SA和MeJA的响应更为迅速, 其表达量在处理2 h后便达到最高值, 随后出现降低趋势, 与对照相比表达变化差异显著( P<0.05)(图5)。因此, TaNPR1、 TaNPR2和 TaNPR3在SA和MeJA处理后均被诱导表达, 表明上述基因可能参与了信号分子SA和JA介导的抗病信号途径。
| 表2 TaNPR1、TaNPR2和TaNPR3蛋白的理化性质 Table 2 Physical and chemical parameters for TaNPR1, TaNPR2, and TaNPR3 |
qRT-PCR分析表明, 在未接菌时, TaNPR1表达量较低, 而在接种赤霉菌后, 抗病材料Apogee73S2中 TaNPR1被迅速诱导并在12 h出现表达高峰, 其表达量约为Apogee中 TaNPR1表达量的2.58倍( P<0.05), 随后呈现出下降趋势; 与其相比, TaNPR1在感病材料Apogee中表达量虽然也逐渐升高, 但直到48 h才达到最高值。 TaNPR2对赤霉菌的侵染响应较为缓慢, 并且在整个胁迫过程中抗、感材料表达量变化未出现显著差异( P<0.05)。 TaNPR3在赤霉菌诱导处理后24 h内表达量逐渐升高, 并在24 h达到顶峰, 此后的24 h到72 h表达量虽有所下降, 但仍显著高于对照( P<0.05), 而且 TaNPR3在接菌后48 h内的表达量在抗病材料Apogee73S2中显著高于在感病材料Apogee中( P<0.05)(图6)。
首先在拟南芥中通过图位克隆获得 NPR1基 因[5], 随后在多种植物中都发现 NPR1基因及同源基因[16]。作为植物抵抗病原菌胁迫过程中的一个核心元件, NPR1介导的抗性可能在单子叶及双子叶植物中也十分保守[15]。本研究从小麦中分离得到3个 NPR1-like基因的全长 cDNA序列, 序列分析表明, TaNPR1、TaNPR2及TaNPR3具有BTB/POZ和ANK功能域, 这2个功能域在NPR1蛋白中高度保守, 并且参与蛋白质间的相互作用。其中, 突变ANK结构域中的npr1-1和靠近N端的npr1-2氨基酸位点, 会造成NPR1不能与TGA转录因子结合, 导致 PR基因诱导失败并使植物更容易感病[23]。在C端突变nim1-4位点并不影响NPR1与TGA转录因子的结合, 却也会造成SAR反应不能被正常诱导[15], 推测nim1-4突变可能影响NPR1与其他SAR信号蛋白的结合。多重序列比对结果显示, TaNPR1、TaNPR2及TaNPR3在npr1-1、npr1-2和nim1-4氨基酸位点
 | 图5 SA和MeJA诱导下 TaNPR1(A)、 TaNPR2(B)和 TaNPR3(C)的表达Fig. 5 Expression of TaNPR1(A), TaNPR2(B), and TaNPR3(C) induced by SA and MeJA |
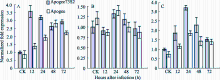 | 图6 赤霉菌诱导下 TaNPR1(A)、 TaNPR2(B)和 TaNPR3(C)的表达Fig. 6 Expression of TaNPR1(A), TaNPR2(B), and TaNPR3(C) induced by F. graminearum |
十分保守。在正常状态下, NPR1能够通过二硫键形成寡聚体存在于细胞质中, 而保守的半胱氨酸残基对于二硫键的形成十分必要。在拟南芥中, 半胱氨酸残基Cys82和Cys216对于NPR1寡聚体的形成至关重要[8]。而在水稻中也存在这2个保守的半胱氨酸, 分别位于第83和第211位[24]。本研究对TaNPR1、TaNPR2和TaNPR3氨基酸序列分析发现, TaNPR1同样存在2个保守的半胱氨酸活性位点, 分别为Cys71和Cys211。但TaNPR2与TaNPR3仅在第一个半胱氨酸位点处保守, 另一个位点被L代替, 在拟南芥、水稻的NPR1同源物AtNPR3、AtNPR4和OsNPR2、OsNPR3中也同样如此。表明NPR1同源基因虽然在该活性位点与NPR1存在差异, 但在不同物种中仍十分保守。此外, 3个小麦NPR1-like蛋白还具有NPR1蛋白家族代表性的NPR1_like_C结构域。综合以上结果可以看出, TaNPR1、TaNPR2及TaNPR3在结构上的保守性对行使抗病功能具有十分重要意义, 但其在小麦抗病反应中所起到的具体作用尚需进一步验证。
进化树分析显示, 小麦TaNPR1、TaNPR2和TaNPR3分别与单子叶植物的NPR1、NPR2及NPR3进化关系较近, 其中与大麦的NPR蛋白关系最近, 推测在进化过程中, TaNPR1、TaNPR2和TaNPR3可能分别与大麦HvNPR1、HvNPR2和HvNPR3存在直向同源关系。TaNPR1与TaNPR2、TaNPR3在进化树中分属不同分支, 同源关系较远。TaNPR1与AtNPR1及其他物种的NPR1蛋白关系较近, 而TaNPR2和TaNPR3则与包括AtNPR3、AtNPR4在内的NPR1同源蛋白的进化关系较近, 表明TaNPR2和TaNPR3可能为TaNPR1的旁系同源物。尽管SA诱导的SAR反应需要调控因子NPR1的参与, 但
NPR1并不直接与SA结合。最近在拟南芥中的研究表明, NPR1的同源物NPR3和NPR4是水杨酸的受体, 能够与其结合并作为泛素E3连接酶Cullin 3的转接蛋白(adaptor)介导NPR1降解[19]。在拟南芥 npr3/npr4双突变体中虽然累积了较多的NPR1, 但对SAR反应并不敏感[19]。这表明NPR3和NPR4在生物学功能上与NPR1不同, 可以通过与SA的结合对NPR1介导的抗病反应起到十分重要的调控作用。目前水杨酸介导的小麦抗病网络并不明确, 而在小麦中 TaNPR1、 TaNPR2、 TaNPR3是否分别行使与 AtNPR1、 AtNPR3和 AtNPR4类似的功能, 有待深入研究。
一般认为, SA介导的SAR反应主要对活体营养型病菌起作用, 而JA/ET介导的抗病信号途径对抵抗腐生型真菌的侵染起重要作用[25]。引发小麦赤霉病的禾谷镰刀菌是一种半腐生型真菌, 因而小麦赤霉病抗病机制可能更加复杂。本研究通过荧光定量PCR分析了SA、MeJA处理及赤霉菌诱导下3个小麦 NPR1-like基因的表达情况, 发现SA、MeJA均能明显诱导 TaNPR1及其同源基因 TaNPR2和 TaNPR3的表达。在接种赤霉菌后, 虽然抗、感近等基因系中 TaNPR1和 TaNPR3都能响应赤霉菌的诱导而上调表达, 但在抗病材料Apogee73S2中这2个基因在接种后的响应更为迅速、表达量更高。与 TaNPR1和 TaNPR3相比, TaNPR2在赤霉菌诱导后, 感、抗材料中均未出现显著差异。因此推测抗病材料Apogee 73S2中, TaNPR1和 TaNPR3对赤霉菌侵染前期的快速响应可能对赤霉病抗性起重要作用, 而 TaNPR2无显著作用。本研究中 TaNPR1、TaNPR2及 TaNPR3基因的克隆, 为进一步研究 NPR1-like基因在小麦赤霉病抗病反应中的作用以及构建小麦水杨酸、茉莉酸路径调控网络奠定了基础。
从小麦抗赤霉病近等基因系中克隆获得3个与拟南芥 AtNPR1结构类似的 NPR1-like基因, 分别命名为 TaNPR1、 TaNPR2和 TaNPR3, 其编码的3个NPR1-like蛋白中, TaNPR1的同源性与AtNPR1最高, 而TaNPR2、TaNPR3与AtNPR3、AtNPR4同源关系较近。 TaNPR1和 TaNPR3不但能被SA和MeJA所诱导, 还能快速响应赤霉菌的侵染而明显上调表达; 而 TaNPR2虽然也能响应SA和MeJA的诱导, 但对赤霉菌的侵染并无显著响应。因此, TaNPR1和 TaNPR3可能参与了小麦赤霉病的抗病防御过程。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|